下图为三种常用的模型构建策略路线图I、II、III。

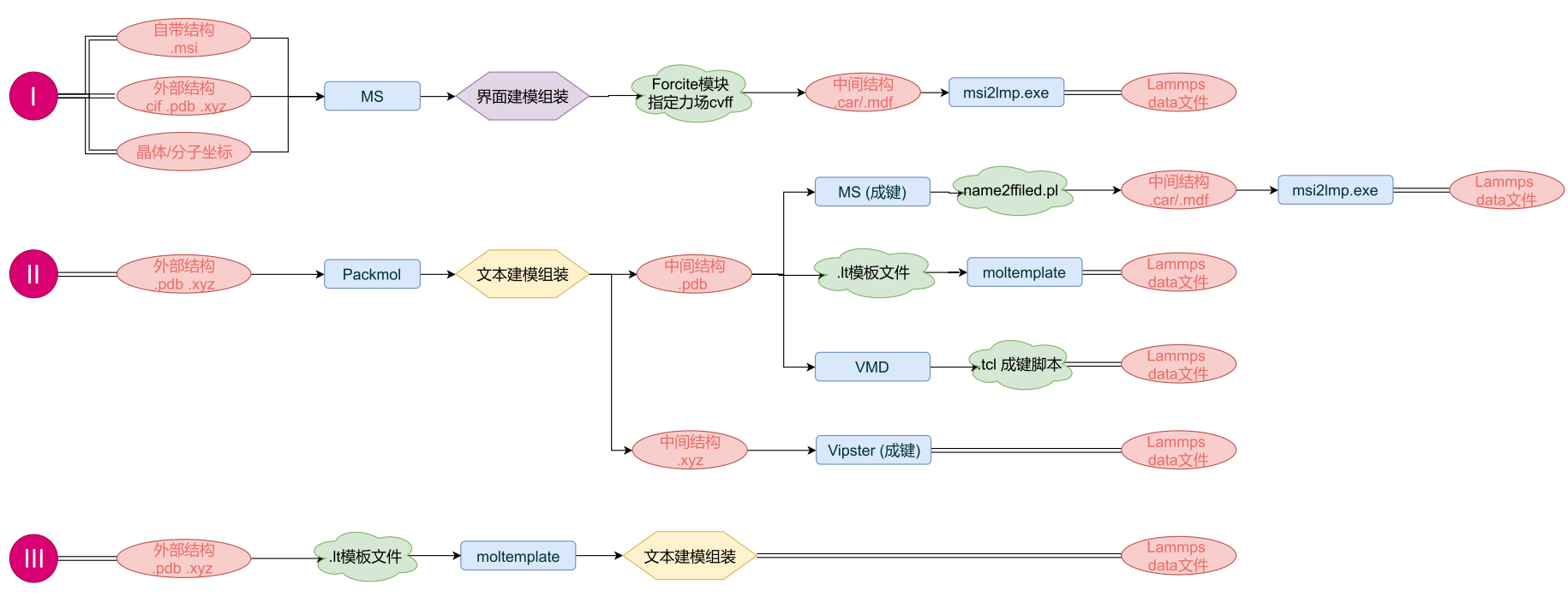
通常情况下路线I能完成许多基本的模型构建和导出,当遇到一些较复杂的模型需要组装时就会显得力不从心了。如下图这种,多组分固液模型组装。

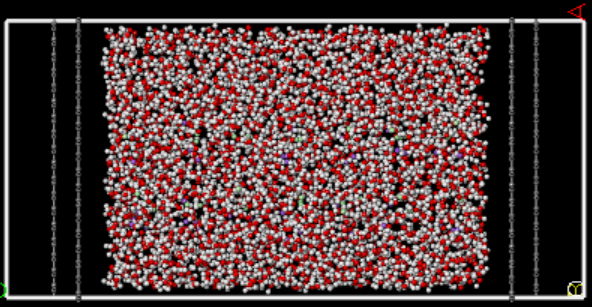
此时,我们需要利用到组装工具软件Packmol,按照路线II完成组装和转化。
在
MS中构建好所需要的所有组分结构,导出为.pdb文件。

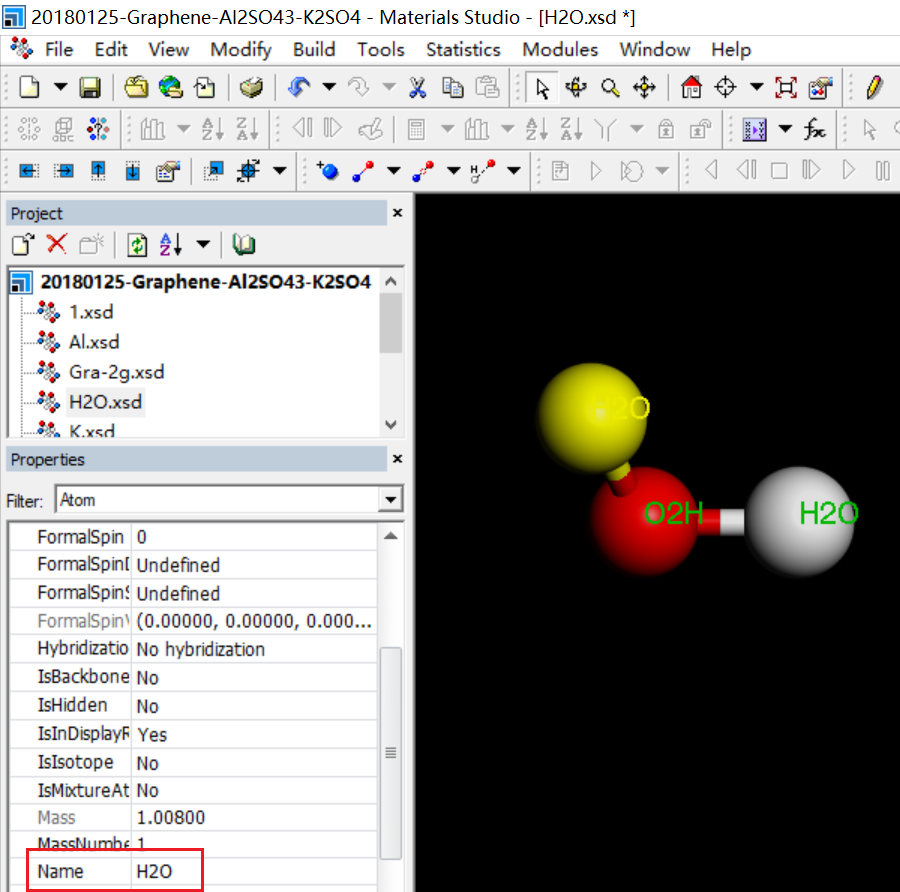
需要注意的是,导出前需要手动设置每个原子的Name,相同原子类型的设置为相同Name,不同原子类型需要设置为不同Name,此处设置为该路线中的关键步骤。

编写用于
Packmol组装的in文件,利用packmol.exe可执行程序完成组装,得到组装完成结构文件structure_Gra_Al2SO4_3-0.6M.pdb。
# 距离容差, 不同分子中原子间的最小距离2A tolerance 2.0 # 输入输出文件的格式 filetype pdb # 输出文件的名字 output structure_Gra_Al2SO4_3-0.6M.pdb # structure Gra.pdb number 1 center fixed 19.0179 18.45 30.01 0. 0. 0. end structure # structure H2O.pdb number 2840 inside box 1.0 1.0 1.0 37.5 37.0 59.0 end structure # structure Al.pdb number 60 inside box 1.0 1.0 1.0 37.5 37.0 59.0 end structure # structure SO4.pdb number 90 inside box 1.0 1.0 1.0 37.5 37.0 59.0 end structure
将组装完成结构文件
structure_Gra_Al2SO4_3-0.6M.pdb重新拖回到MS中。任意选中一个原子,可以发现原子属性中Name栏为预设原子Name,而ForcefiledType力场类型栏为空。

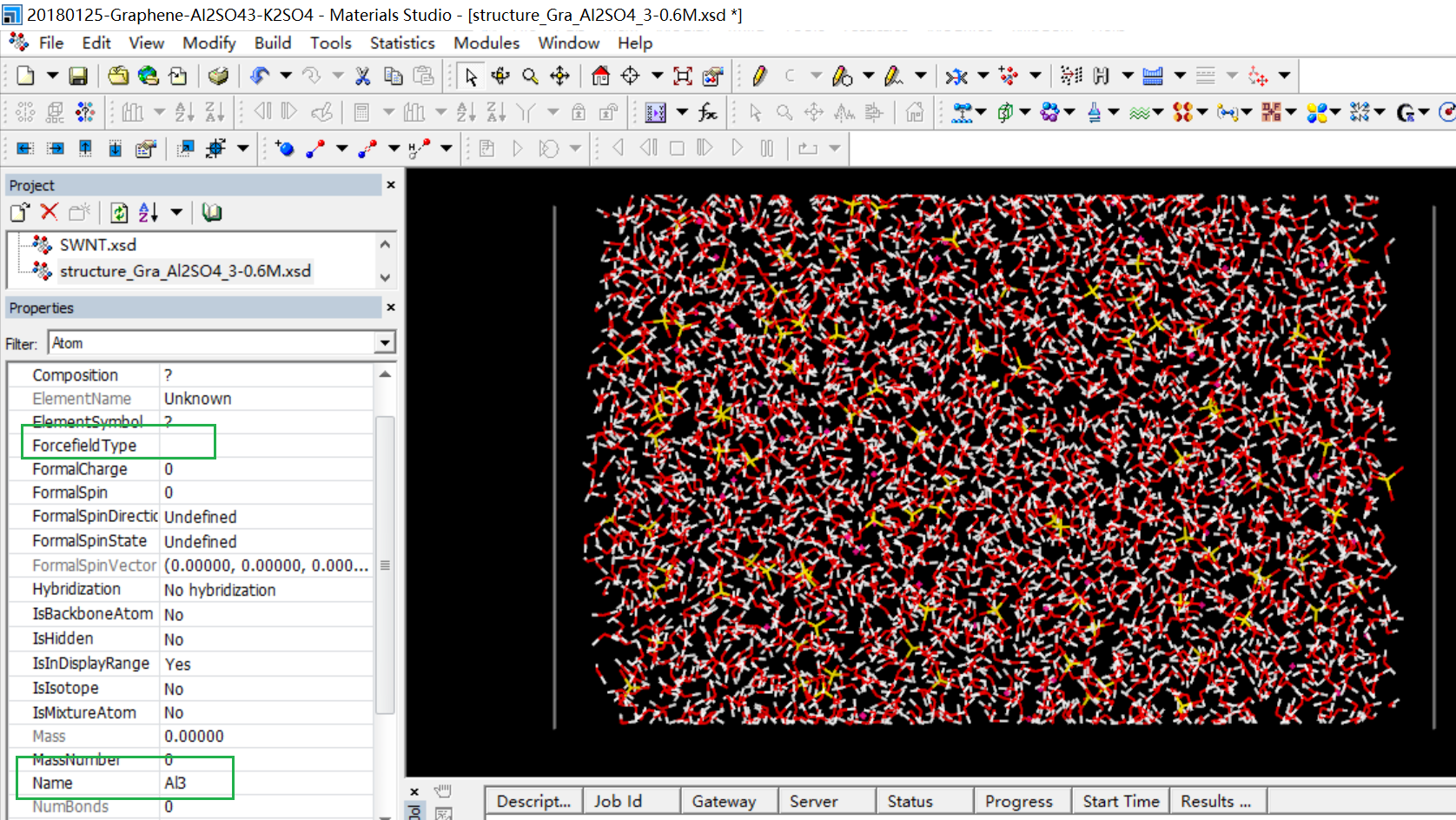
此时我们借助name2ffield.pl脚本将结构中所有原子Name属性复制到ForcefiledType属性。

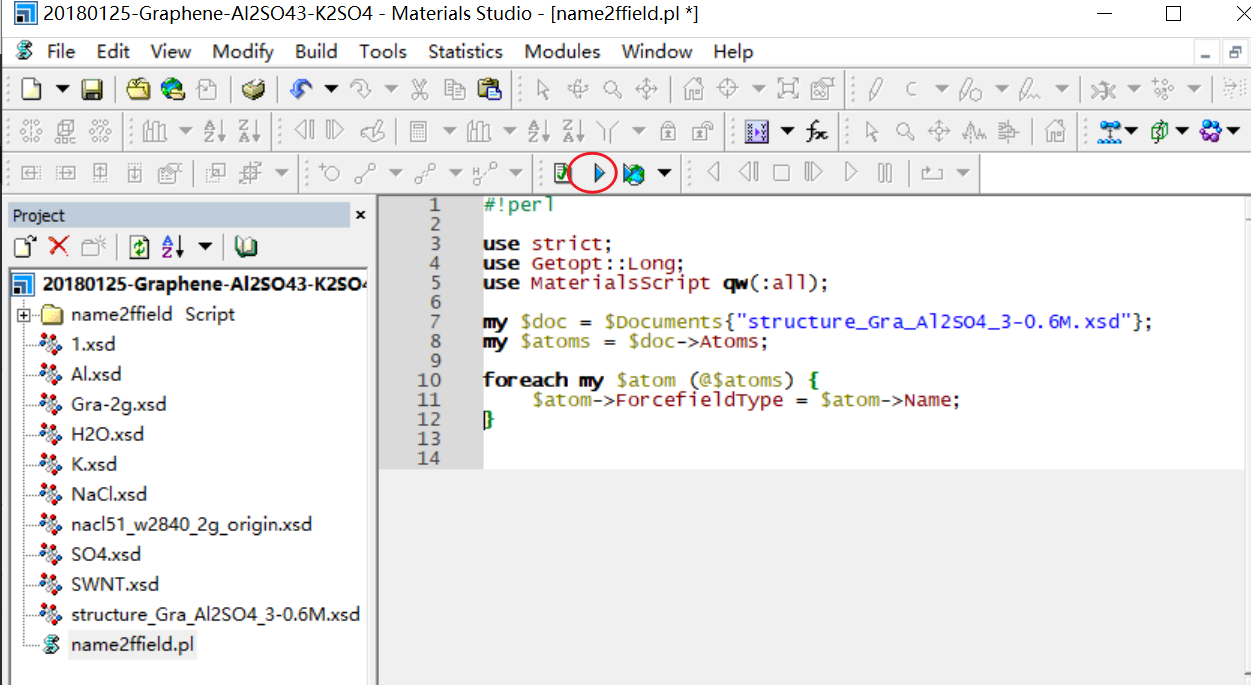
name2ffield.pl文件内容如下
#!perl
use strict;
use Getopt::Long;
use MaterialsScript qw(:all);
my $doc = $Documents{"structure_Gra_Al2SO4_3-0.6M.xsd"};
my $atoms = $doc->Atoms;
foreach my $atom (@$atoms) {
$atom->ForcefieldType = $atom->Name;
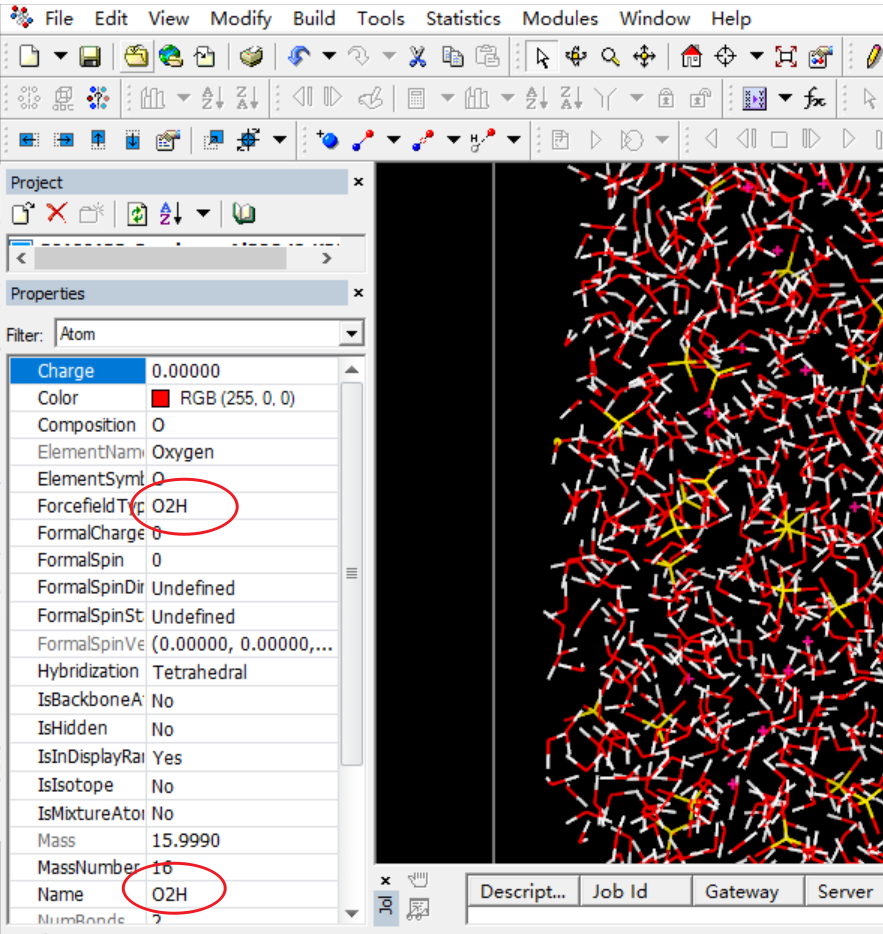
}name2ffield.pl脚本执行完,可以随意选择其中几个原子检查ForcefiledType力场类型设置是否正确完成。

ForcefiledType力场类型设置完成后,在MS中添加晶格。

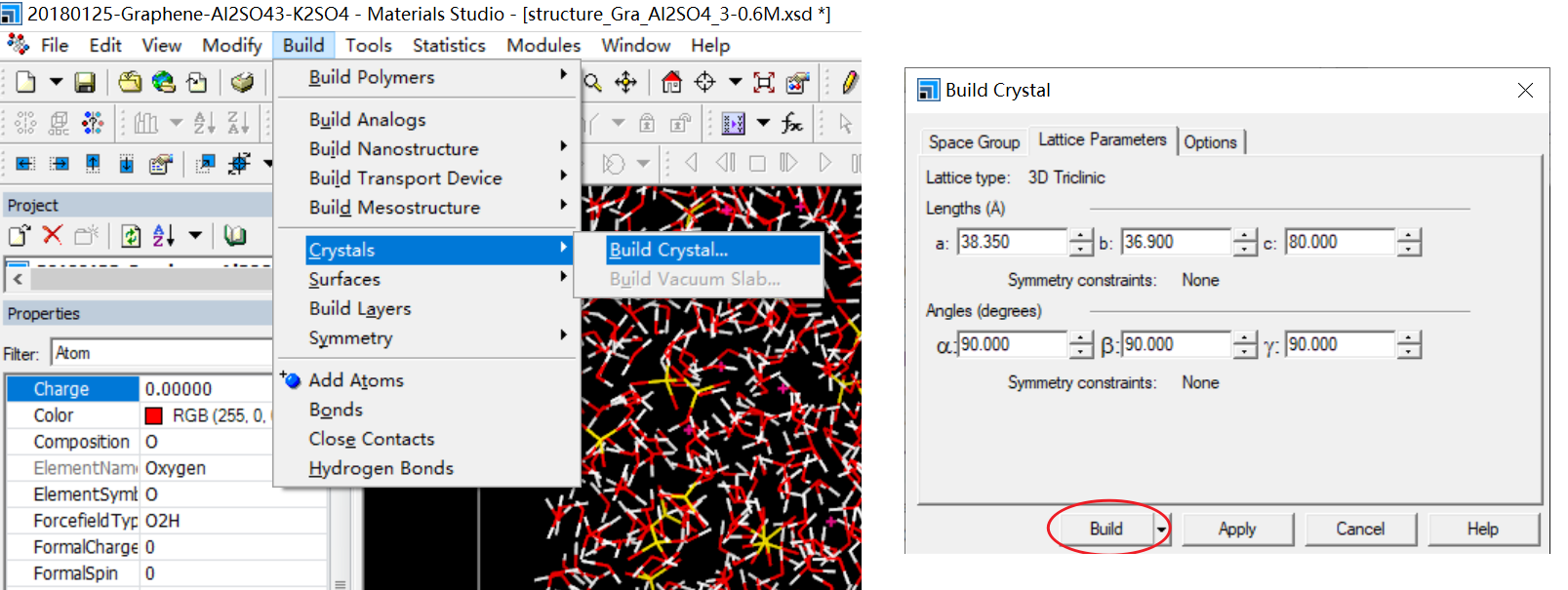
晶格添加完成后模型

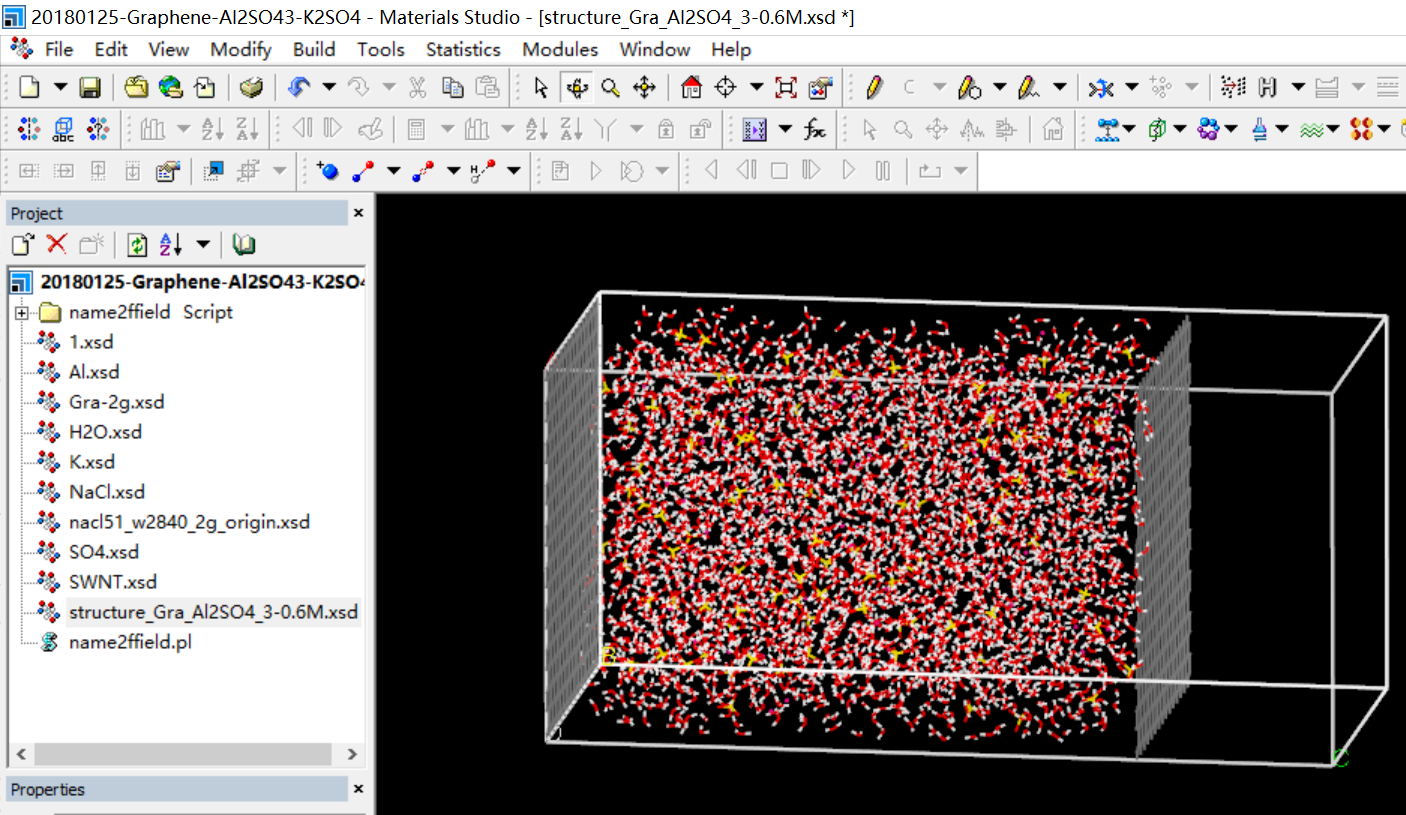
导出为为
.car文件格式,然后利用msi2lmp.exe小程序来完成格式的转换,即可生成需要的data文件structure_Gra_Al2SO4_3-0.6M.data。重复路线I中对应检查。使用Ovito软件查看data文件,可得如下所示结构。

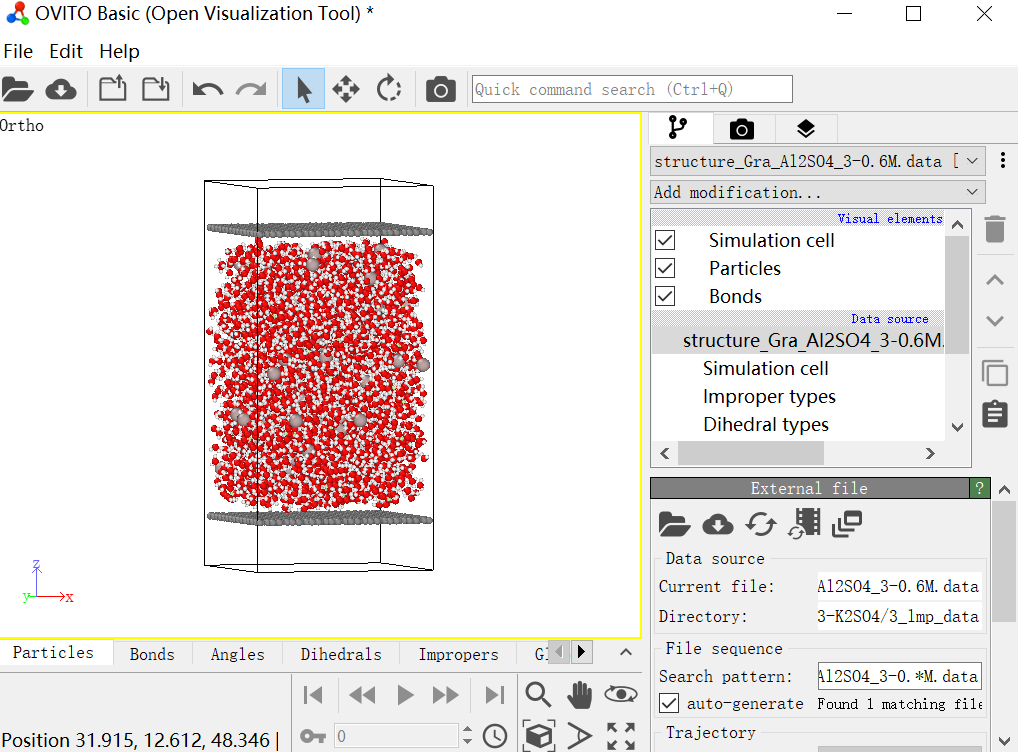
附件:本案例中对应结构文件
![]() 20180125-Graphene-Al2SO43-K2SO4.7z
20180125-Graphene-Al2SO43-K2SO4.7z
