### 在计算模拟中,经常会遇到需要对纯相原子结构进行随机掺杂的情况。今天给大家介绍一个巧用`Matlab`脚本实现该目的
- 软件需要 `MS`,`Matlab`
1. 在`MS`中建立原始结构模型,此处为石墨烯为例,结构模型如下图所示,模型中只含有C原子,以`pdb`格式导出结构文件,文件名为`gra-ort-min-sc.pdb`

2. 如果希望在上述模型中掺入10%的B和10%N原子,掺杂位置随机,该如何实现?
答:可以通过在`Matlab`软件中运行下面`Matlab`脚本实现。
%% 元素掺杂clc; clear; origin_data_file = 'gra-ort-min-sc.pdb'; % 原始pdb文件名
num_atom_number = 880; % 总原子个数
name_replace_atom = 'C'; % 被替换原子名称
name_new_atom = ["N","B"]; % 替换原子名称
rtio_new_atom = [0.1, 0.1]; % 替换原子比例
num_pdbdata_head = 9; % 文件头行数
fid_pdbdata = fopen(origin_data_file,'r'); % 打开原始pdb文件
data_pdbdata_head = textscan(fid_pdbdata,'%s',num_pdbdata_head,'delimiter','\n'); % 读入文件头信息
data_pdbdata_xyz = textscan(fid_pdbdata,'%s %f %s %s %f %f %f %f %f %f %s \r\n',num_atom_number); %读入坐标信息
data_pdbdata_tail = textscan(fid_pdbdata,'%s','delimiter','\n'); % 读入文件尾信息fclose(fid_pdbdata); %关闭原始pdb文件
[rep_atom_position, ~] = find(strcmp(data_pdbdata_xyz{1,3},name_replace_atom)); % 找到被替换原子在data_pdbdata_xyz矩阵中所在行
[n_all_atoms, ~] = size(data_pdbdata_xyz{1,3}); % 确定总原子个数
[n_rep_atoms, ~] = size(rep_atom_position); % 确定总的可替换原子个数
[~, doping_type] = size(rtio_new_atom); % 确定替换原子种类
r_random_index = randperm(n_rep_atoms)'; % 生成大小为总的可替换原子个数的随机矩阵
index_start = 1; % 随机矩阵起始值
for i_doping_type = 1:doping_type % 对多种替换原子种类进行替换 i_random_num = fix(rtio_new_atom(1,i_doping_type) * n_rep_atoms); % 计算替换原子个数 i_random_index = r_random_index(index_start:i_random_num+index_start-1); % 在随机矩阵中中取对应个数原子 index_start = index_start + i_random_num; % 随机矩阵起始值递进 for i_replace_index = 1:i_random_num % 对替换原子个数进行替换 replace_index = i_random_index(i_replace_index); % 确定替换原子在rep_atom_position矩阵中行数 replace_position = rep_atom_position(replace_index,1); % 确定替换原子在data_pdbdata_xyz矩阵中行数 data_pdbdata_xyz{1, 3}{replace_position, 1} = name_new_atom(1,i_doping_type); % 替换第三列原子名 data_pdbdata_xyz{1,11}{replace_position, 1} = name_new_atom(1,i_doping_type); % 替换第十一列原子名 end endfid_pdbdata_new = fopen(strcat('Doped_',origin_data_file),'w+'); % 新建替换后pdb文件for i = 1:length(data_pdbdata_head{1, 1}) % 写入文件头信息 fprintf(fid_pdbdata_new,'%s\n',data_pdbdata_head{1, 1}{i, 1});endfor i = 1:length(data_pdbdata_xyz{1, 1}) % 写入坐标信息 fprintf(fid_pdbdata_new,'%4s', data_pdbdata_xyz{1,1}{i,1}); fprintf(fid_pdbdata_new,'%7.0f',data_pdbdata_xyz{1,2}(i,1)); fprintf(fid_pdbdata_new,'%3s', data_pdbdata_xyz{1,3}{i,1}); fprintf(fid_pdbdata_new,'%6s', data_pdbdata_xyz{1,4}{i,1}); fprintf(fid_pdbdata_new,'%6.0f',data_pdbdata_xyz{1,5}(i,1)); fprintf(fid_pdbdata_new,'%12.3f',data_pdbdata_xyz{1,6}(i,1)); fprintf(fid_pdbdata_new,'%8.3f',data_pdbdata_xyz{1,7}(i,1)); fprintf(fid_pdbdata_new,'%8.3f',data_pdbdata_xyz{1,8}(i,1)); fprintf(fid_pdbdata_new,'%6.2f',data_pdbdata_xyz{1,9}(i,1)); fprintf(fid_pdbdata_new,'%6.2f',data_pdbdata_xyz{1,10}(i,1)); fprintf(fid_pdbdata_new,'%12s \n',data_pdbdata_xyz{1,11}{i,1});endfor i = 1:length(data_pdbdata_tail{1, 1}) % 写入文件尾信息 fprintf(fid_pdbdata_new,'%s\n',data_pdbdata_tail{1, 1}{i, 1});end fclose(fid_pdbdata_new);
3. 运行脚本后得到文件名为`Doped_gra-ort-min-sc.pdb`的掺杂后结构文件,将此`pdb`文件拖入到`MS`中,得到原子结构如下
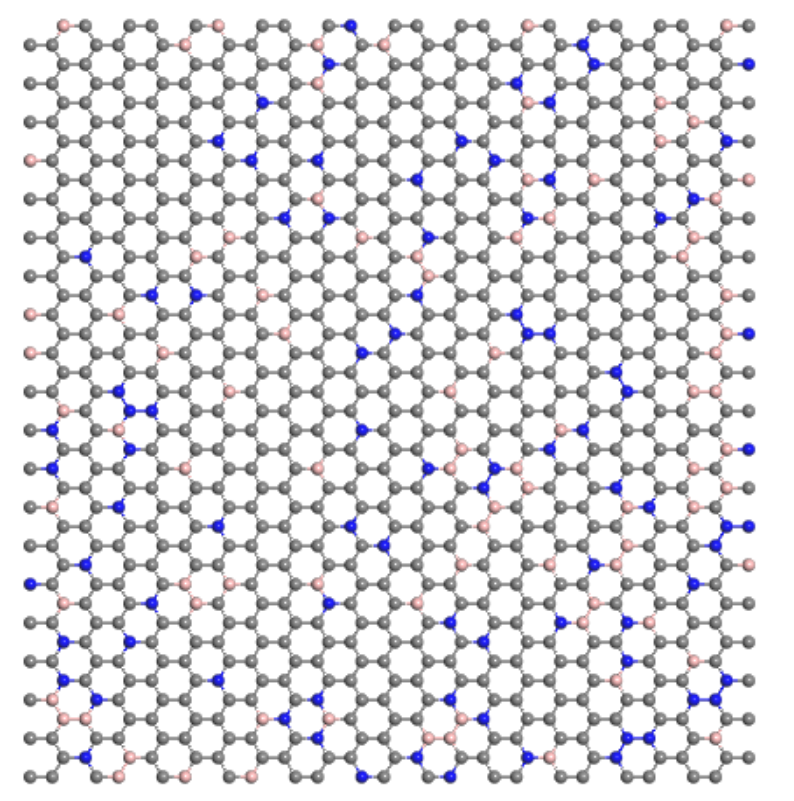
4. 在不同实际使用情况中,可对应调整5-10行等号后内容,以实现具体掺杂目标,如改变掺杂原子,改变掺杂比例等。
- 课后练习:如果只想掺杂10% B原子中,该如果修改源代码实现此目标?
### 本案例中涉及所有脚本和结构文件附件:
